Research on the Algorithm of Mining Information of Traditional Chinese Herb System Biology Based on Graph Neural Network
-
摘要:目的
构建中药-基因-蛋白复杂网络,优化中药潜在关联基因的挖掘方法,提升中药系统生物学信息的挖掘效能,为进一步探究中药作用机制提供帮助。
方法提出融合注意力机制的图神经网络模型HERBGAT,以公开数据平台中少量的中药关联基因数据为输入,在中药-基因-蛋白复杂网络中进行深度挖掘,输出潜在的中药关联基因,将预测结果通过生信平台进行Disease关联分析、KEGG信号通路分析阐明其作用机制,并借助文献检索平台进行预测结果验证。
结果训练结果表明,HERBGAT模型预测准确率均值可达94%,相较于其他2种先进的复杂网络挖掘方法,HERBGAT在ACC、AUC和AUPR三项指标中均表现出更优秀的性能;在文献验证环节,模型预测结果得到中医临床文献及现代药理学文献证明,展现出HERBGAT在实际应用中的良好效果。最后,以借助HERBGAT模型和改进的EMOGI模型探究半夏治疗肺癌作用机制为例,发现半夏治疗肺癌的潜在关联基因199个,并借助生物信息学方法对这些潜在关联基因进行初步分析探讨。
结论HERBGAT模型能有效挖掘潜在的中药关联基因,提高中药-基因-蛋白复杂网络的挖掘效能,为中药系统生物学信息挖掘方法的优化提供新的思路与参考,为探究中药作用机制等研究提供数据基础及实验方向。
Abstract:OBJECTIVETo provide help for further exploring the mechanism of action of traditional Chinese herb by constructing a complex network of traditional Chinese herb-gene-protein, optimizing the mining method of potential associated genes of traditional Chinese herb and improving the mining efficiency of traditional Chinese herb system biology information.
METHODSA graph neural network model HERBGAT with an attention mechanism was proposed. A small amount of traditional Chinese herb-related gene data in the public data platform was used as input, and deep mining was performed in the traditional Chinese herb-gene-protein complex network to output potential traditional Chinese herb-related genes. The prediction results were analyzed by disease association analysis and KEGG signaling pathway analysis on the bioinformatics platform to clarify their mechanism of action, and the prediction results were verified by the literature retrieval platform.
RESULTSThe training results showed that the average prediction accuracy of the HERBGAT model could reach 94%. Compared with the other two advanced complex network mining methods, HERBGAT showed better performance in the three indicators of ACC, AUC and AUPR. In the literature verification stage, the model prediction results were verified by TCM clinical literature and modern pharmacology literature, showing the good effect of HERBGAT in practical application. At the end of this paper, taking the HERBGAT model and the improved EMOGI model to explore the mechanism of action of Pinellia ternata in treating lung cancer as an example, 199 potential associated genes of Pinellia ternata in treating lung cancer were found, and these potential associated genes were preliminarily analyzed and discussed with the help of bioinformatics methods.
CONCLUSIONThe HERBGAT model can effectively mine potential traditional Chinese herb-associated genes, improve the mining efficiency of traditional Chinese herb-gene-protein complex networks, provide new ideas and references for the optimization of traditional Chinese herb system biology information mining methods, and provide data basis and experimental direction for exploring the mechanism of action of traditional Chinese herb.
-
中药是中医防病治病的物质基础[1],其以“君臣佐使”为用药规律,注重药物的配伍联合使用,而中药关联的靶点基因是研究中药作用机制的关键之一。随人工智能时代到来,复杂网络在系统生物学的应用越来越广泛,特别是图神经网络的算法在相关领域也取得重要研究进展。图神经网络算法能够协助科研人员利用海量公开的生物医学数据,深入分析研究中药的作用机制。程建超等[2]使用关联规则分析肿瘤标志物与核心中药之间的关联,发现白花蛇舌草、半枝莲与肿瘤标志物指标的改善关联度较高。周雪忠等[3]通过建立网络医学框架,成功通过中药靶点和症状模块的网络相似程度,预测中药治疗症状的有效性。Mastropietro等[4]通过图神经网络的方法,通过分析蛋白质与配体相互作用的图表示结构成功预测配体的亲和力。所以,利用复杂网络结合新兴的图神经网络算法,可以帮助科研人员有效整合系统生物学数据,并深入挖掘出中药-基因-蛋白复杂网络中隐含的有价值信息,为新的中药靶点基因预测和中药作用机制的探究提供便利。
本研究以系统生物学多组学数据为基础,构建包含多组学数据的中药-基因-蛋白复杂网络,利用图神经网络分析方法,对中药潜在关联基因进行挖掘与发现;借助生信平台,利用挖掘出的中药潜在关联基因,从系统生物学视角探寻、分析这些基因的关联疾病及中药对疾病的作用机制;从国医大师周仲瑛治疗肺癌的核心药物中选取半夏为例,对半夏治疗肺癌的关键靶点基因、关键信号通路进行识别及分析,从系统生物学的角度,结合中药多靶点、多作用途径的药效作用特点,探寻半夏的核心作用机制。最后,本研究检索大量中药类临床文献、药理学分析文献,对挖掘出的系统生物学信息进行文献验证及补充说明。综上所述,本研究提出融合注意力机制的图神经网络模型HERBGAT,进行中药系统生物学信息的挖掘,以期为潜在中药关联基因的挖掘方法提供新的思路与参考,为探究中药作用机制的相关研究提供帮助。
1. 方法
1.1 数据来源
1.1.1 中药化合物选取及收集
本研究从中药数据库入手,结合各大医学数据库收录文献与中医古籍中筛选得到的候选中药活性成分。依托TCMSP[5]等中医药病证数据库收集中药化合物;接着在CNKI、PubMed[6]等文献数据库中检索有关中药活性成分及诊疗处方的文献,对中药化合物进行进一步筛选和交互验证。
1.1.2 基因和蛋白质组学数据获取
本研究采用的疾病关联基因数据主要源于TCGA[7]、HGNC[8]、OMIM、StringDB、GeneCards等相关的人类遗传病数据库、蛋白质网络数据库等系统生物学数据库,在其中提取出本研究需要的疾病关联基因,并将多组学基因特征纳入基因节点当中;采用的中药关联基因数据主要来自PubChem[9]数据库或HIT数据库[10],用于完成中药-基因-蛋白复杂网络的构建。
1.2 研究方法
1.2.1 多组学癌症信息纳入复杂网络
针对多组学数据的整合提取,本研究主要选取癌症样本,通过在公开数据平台中,收集海量正常样本与癌症样本数据,比较样本间差异性获得多组学数据。首先在TCGA中下载关于12种癌症的29 446组样例用于基因差异表达、突变和DNA甲基化的基因特征提取与多组学特征基因蛋白网络构建。在从ConsensusPathDB[11]中下载的蛋白质相互作用(PPI)网络设置combined_score>850进行边的初次筛选,并将筛选后的边进行去重、边分数排序等处理,将处理好的蛋白质网络转为边连接矩阵和节点名称矩阵,其中本研究计算了3个基因的生物学特征指标纳入复杂网络中,参与多组学特征基因蛋白网络的构建,包括基因差异表达率、基因差异DNA甲基化率和基因突变率。
基因差异表达率的评价指标是基因差异表达倍数(Fold changes, FC),每个基因通过其在癌症与匹配的正常样本中的表达值之间的log2倍变化来测量,具体见公式(1)。其中FCc代表基因c的差异表达倍数, median(Pc)代表基因c在所有癌症样本中的表达值的中位数,median(Nt)代表基因c在所有正常样本中的表达值的中位数。
$$ F C_c=\log \left[\frac{{median}\left(P_c\right)}{{median}\left(N_t\right)}\right] $$ (1) 基因DNA甲基化率数据中,从TCGA数据库中收集肿瘤和邻近正常组织的DNA甲基化数据。收集12种癌症总计4 635个癌症样例,经过批次矫正[12],对每种癌症的癌症样本和正常样本进行规范化处理,针对每种癌症,求出各个基因对应的平均甲基化水平值。
基因突变率包括拷贝数变异(Copy number variations, CNV)和单核苷酸位点变异(Single nucleotide variants, SNV)。基于TCGA数据库,针对CNV数据,本研究根据基因长度比例,求出每种癌症对应基因的平均值。针对SNV数据,按照HotNet2[13]的数据处理方法进行前期处理。最终汇总整合12种癌症总计4 859个样例,将每种癌症对应基因的SNV值与CNV平均值整合生成对应特征矩阵。
最后,将12种癌症类型中每个基因的生物学特征串联起来,并进行min-max正常化。至此,本基因蛋白网络中每个基因节点都纳入了一个36维的生物特征载体,由12个突变率值、12个甲基化值和12个基因表达值组成。接下来,继续在此基因蛋白复杂网络中纳入中药相关数据,进行阳性、阴性样本的标识,并最终完成中药-基因-蛋白复杂网络的构建工作。
1.2.2 中药-基因-蛋白复杂网络构建
复杂网络最早起源于图论,在经历了一段时间的发展后,理论体系和结构初具雏形。在1998年6月与1999年10月由Watts[14]与Barabasi[15]两位学者先后提出了复杂网络的相关基本概念,开启了复杂网络研究的新纪元。生物复杂网络的构建以基础蛋白网络,多组学数据为主,结合生物信息学分析技术,辅以药物靶点数据、文献数据等加以补充和验证,可以有效处理分析存储于公开数据平台上的海量药物靶点及基因蛋白数据。
本研究借助TCMSP、PubChem及HIT数据库,通过两种途径获取中药关联基因,一是通过活性成分,在PubChem数据库中,通过获取活性成分关联基因间接提取,二是在基因靶点数据库中直接提取。将这些靶点基因信息纳入复杂网络当中,为复杂网络中已知的中药关联基因节点进行属性标识,为构建中药-基因-蛋白复杂网络奠定了中药关联信息的数据基础。
中药-基因-蛋白复杂网络的构建,以基因为桥梁,在致病基因和中药靶点基因间构建起疾病与中药的关系,为接下来使用图神经网络模型对于关键节点的特征提取及节点预测提供数据网络基础。
1.2.3 HERBGAT模型设计
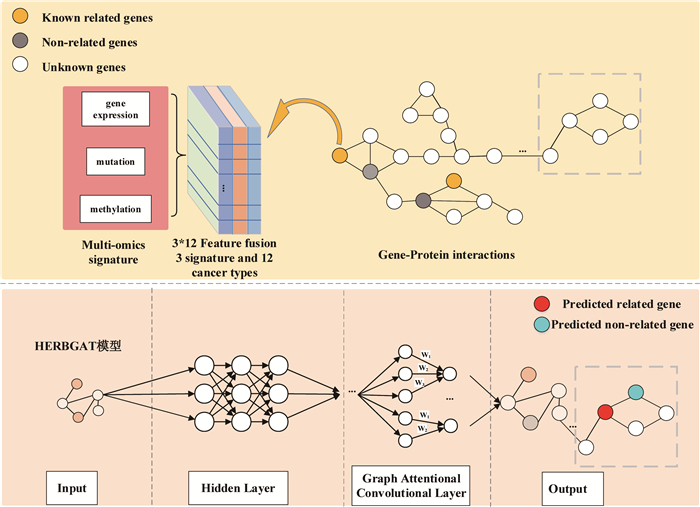
在完成中药-基因-蛋白复杂网络构建后,需要有效提取出中药关联基因的节点共有特征并充分解析节点间关联特性,从节点特征与网络结构特征两个方面,对潜在的、属性未知的基因节点进行分类预测。在中药系统生物学信息挖掘领域,以往的潜在关联基因挖掘方法包括基于随机游走的PageRank算法[16],基于节点特征的PCA主成分分析算法[17],基于深度学习的GCN图卷积神经网络算法[18]等。这些传统方法已在图节点分类预测分析中取得一定成果[19],但仍具有局限性,如PageRank算法在随机游走过程中注重全局信息,仅考虑节点的连接关系,易忽略节点本身的特征信息;PCA算法偏向线性分析,对异常值敏感且无法充分利用标签信息;GCN算法对邻居节点的聚合较为固定,易忽略不同节点的重要性差异。为弥补上述算法的不足之处,并充分考虑中药起效过程是多成分、多靶点作用、多通路调节的复杂生物学过程,本研究提出融合注意力机制的图神经网络模型HERBGAT用于挖掘潜在的中药关联基因。
模型整体架构如图 1所示,在基础的PPI网络图中,白色节点代表基因,具有关联关系的基因节点通过无向边进行联系。通过“1.2.1”中的多组学特征提取工作,本研究将多组学生物特征纳入基因节点结构当中,丰富节点信息,为模型挖掘提供更多有效内容。通过“1.2.2”中的网络构建工作,本研究将指定中药的关联基因在PPI网络中全部标识为黄色节点,将经过筛选后无关联的基因标识为灰色节点,通过对节点给予标签信息的方式,进行半监督学习。
HERBGAT模型基于PyTorch深度学习框架并引入图注意力机制[20],利用DGL库[21]中提供的方法进行图构建及运算。网络结构包括输入层、隐藏层、图注意力卷积层及线性层。模型训练过程如图 1所示,首先将中药-基因-蛋白复杂网络封装在多个特征矩阵输入模型中,其中将全部数据划分为75%的训练集和25%的训练集。接下来在模型初始化过程中,设置随机数种子,将数据打散和随机分配,并加载为小批次样本;初始化Adam参数优化器,负责更新模型所有可学习参数,以最小化损失函数。在模型运算过程中,输入特征通过图卷积神经网络运算经初步处理映射到隐藏层,再经ReLU()激活函数处理后,进入图注意力卷积层,进一步运算并学习到更高层次的节点表示,最终通过线性层映射到最终的输出空间,给出节点的二分类结果。在此模型训练过程中,每个卷积层对复杂网络中进行节点、边特征提取及邻近节点特征聚合,通过层层迭代,在网络全局评估每个节点的关联强度,之后对局部图进行特征提取,并将节点特征归一化处理后,输出至下个网络层。这可以在提取更多局部信息的基础上,识别出其中的高影响力特征及节点,提高模型预测的准确率。
模型中节点信息传播执行多头注意力机制,传播过程中仅计算某节点及其周围一阶邻居之间的结构信息,为不同节点、邻域分配不同注意力系数,最后归一化注意力系数,并将注意力系数权重用来计算节点与其对应特征的线性组合,得到的结果作为每个节点的最终输出特征。在多头注意力机制的运算过程中,每个头在训练过程中自动学习到不同的特征表示,而不是基于人为指定的特征,其可以通过在多个子空间中并行处理信息,允许模型从不同的角度捕捉数据的特征,从而提高模型的性能和泛化能力。具体运算过程为,首先对于每个节点i,计算它所有邻居节点j的未归一化注意力得分eij。这里对于节点i与j的初步得分,本研究使用公式(2)进行计算。其中使用LeakyReLU计算注意力得分,使其即使在负值情况下也能保持一定的梯度,从而避免神经元死亡问题。a为用于计算注意力分数的可学习权重向量,与权重矩阵W变换后的节点特征向量进行点积。hi, hj分别代表节点特征向量。
$$ e_{i j}={LeakyReLU}\left(a^T\left[W h_i \| W h_j\right]\right) $$ (2) 接下来为使不同节点间的系数易于归纳比较,采用softmax函数进行归一化处理,确保每个节点与邻居节点间的注意力系数之和为1,见公式(3)。其中eij代表节点间未归一化注意力得分,经exp函数预处理后,进行归一化处理。
$$ {\alpha _{ij}} = softma{x_j}\left( {{e_{ij}}} \right) = \frac{{exp\left( {{e_{ij}}} \right)}}{{\sum\limits_{k \in {N_i}} {exp} \left( {{e_{ik}}} \right)}} $$ (3) 最终得到注意力机制的计算系数,见公式(4)。其中T表示矩阵转置,‖表示矩阵连接运算。
$$ \alpha_{i j}=\frac{exp \left[{LeakyReLU}\left(a^T\left[W h_i \| W h_j\right]\right)\right]}{\sum\limits_{k \in N_i} exp \left[{LeakyReLU}\left(a^T\left[W h_i \| W h_k\right]\right)\right]} $$ (4) 接下来,将归一化注意力系数αij用于加权邻居节点的特征,从而更新节点的特征表示,见公式(5)。
$$ h_i{ }^{\prime}=\sigma\left(\sum\limits_{j \in N_i \cup\{i\}} \alpha_{i j} W h_j\right) $$ (5) $h_i{ }^{\prime}$是节点i经过更新后的特征向量,σ是激活函数,本研究中采用ReLU()作为激活函数,Ni∪{i}表示节点i及它的邻居节点集合。
通过上述步骤完成模型的构建和训练,本模型可以在中药-基因-蛋白复杂网络中进行高效的特征提取、信息挖掘和节点分类预测。图注意力网络能够有效捕捉节点之间的复杂关系,并为不同节点及其邻域分配不同的权重系数,从而提高模型的性能和鲁棒性。本研究为潜在中药关联基因的发现提供了科学且性能良好的方法支持。
为验证HERBGAT模型的性能,本研究整理选取了公开平台中99味中药的相关数据纳入本研究数据集中,并将HERBGAT模型与其他复杂网络分析算法进行对比,评估每种方法在数据集上的准确率等评价指标。此外,将HERBGAT模型得到的中药潜在基因挖掘结果,进行KEGG等生信分析,并借助中医临床文献和现代药理学文献验证结果的真实有效性。在构建的中药-基因-蛋白复杂网络中,本研究方法与传统复杂网络节点预测分类方法和深度学习图卷积神经网络方法均进行了性能比较。对于传统的机器学习算法,以PageRank算法为代表,对网络中全部基因进行关联性排序,按照排序结果筛选出与已知阳性节点具有高关联性的基因节点,以此估算预测准确率进行性能评价。对于深度学习方法,以基于GCN的HERBGCN方法为代表,通过使用与HERBGAT模型划分相同的训练集、测试集,并给予网络内节点相同的标签信息,同样通过半监督学习的方式计算出模型准确率等相关评价指标进行方法对比。
2. 结果
2.1 多种算法在中药系统生物学信息挖掘中的综合性能评估
HIT数据库中包含中药、中药活性成分和靶点对应的相关信息。为测试模型在中药中的作用效果和模型综合性能,本文在HIT数据库中选取鸡眼草、姜黄、降香、绞股蓝、枸杞子、卷柏、焦槟榔、桔梗、救必应、九节茶、橘红等99味中药在模型中训练,将对应中药及其关联基因纳入模型中进行训练和测试,验证模型对于不同中药条件下的平均性能。
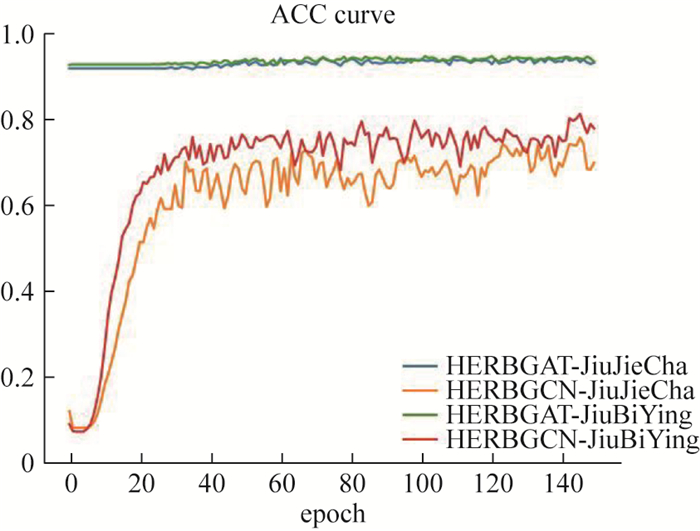
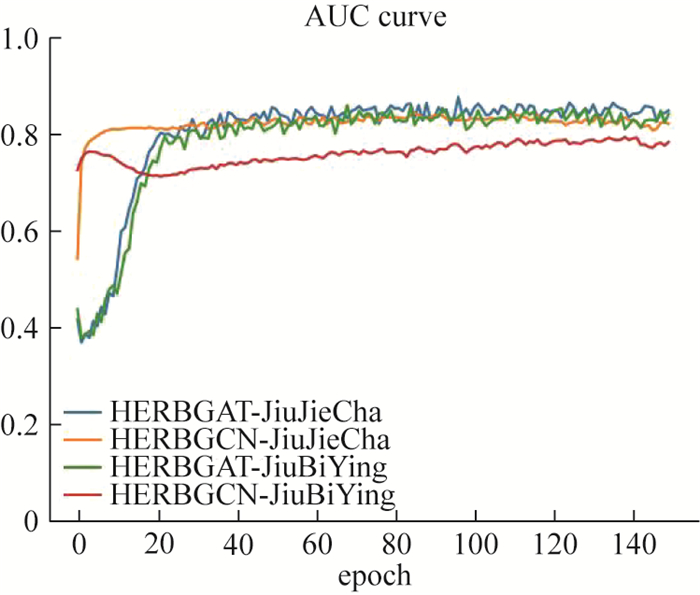
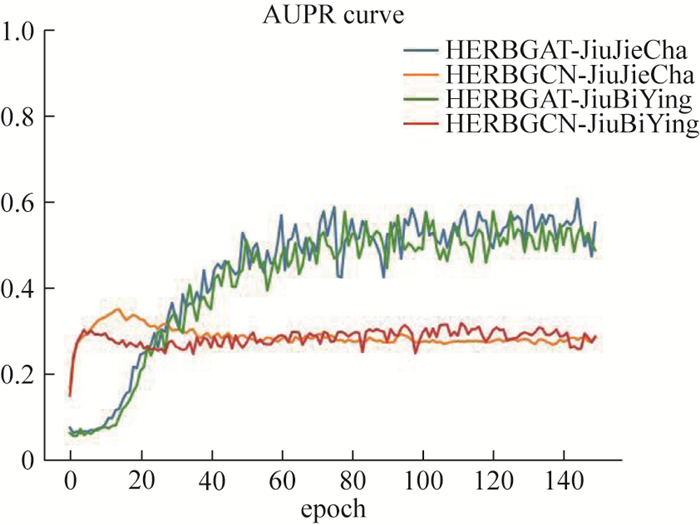
本研究纳入2种图神经网络算法和一种用于处理复杂网络数据的机器学习算法的训练数据进行对比。经过3种算法在99组中药数据集上的模型训练、观测后,在保持复杂网络原有基因特征提取方式不变、整体网络结构不改变的前提下,汇聚中药靶点预测的各项指标信息,求取所有被测数据中三项指标的平均值,这对模型综合性能的评估提供了参考,具体见表 1。在模型运行过程中,本研究观测到模型整体性能与输入的初始中药关联靶点信息的数量有关,在先验基因数量保持在200个至500个时,HERBGAT模型总体性能表现最好,准确率平稳并能维持在94%附近,并且随训练轮次的提高,AUC及AUPR率处于稳步上升状态,模型性能稳定且逐步提高,逐步达到模型运行上限。在HERBGAT算法与HERBGCN算法的训练结果中,选取中药九节茶(JiuJieCha)、救必应(JiuBiYing)绘制训练过程性能曲线图,以此对模型运行过程的性能进行更加直观的展示,见图 2~4。
表 1 3种算法对99种中药训练出的性能均值Table 1. Performance averages of three algorithms trained on 99 traditional herbal medicines算法 ACC AUC AUPR HERBGAT 0.94 0.83 0.52 HERBGCN 0.89 0.70 0.23 PageRank 0.49 0.71 0.12 接下来本文选取8位中药,以疾病为索引,探究预测出的中药潜在关联疾病与现有的中药可产生疗效集间的匹配程度。首先根据3种方法中预测出的每味中药的Top 500基因,首先使用David[22]对其进行疾病富集分析,得到预测出的关联疾病集。接下来,选取中国知网(CNKI)数据库为检索对象,取Top 100的疾病名称为关键词,通过中国生物医学文摘数据库中的主题词、款目词、主题树[23]等多渠道,精确确定疾病检索词,在CNKI中使用全文检索方式,筛选出北大核心文章及SCI文章,检索文献范围包括中医药类临床文献、综述、疾病防治指南、药理学分析等综合性权威文献,得到文献检索结果集后做数据归纳整理。根据预测出的Top 100疾病在文献数据库中的命中率,对算法预测结果进行初步评估,见表 2。结果表明,预测出的中药关联的疾病文献,在大量临床文献、文献综述、药理学分析文献中均取得较高命中率。
表 2 预测的Top100疾病在文献数据库中的检索命中率Table 2. Retrieval hit rates of the predicted top 100 diseases in literature databases中药 HERBGAT_ACC/
%HERBGCN_ACC/
%PageRank_ACC/
%姜 91 93 88 桔梗 89 91 84 白鲜皮 85 82 78 京大戟 68 63 61 橘红 81 81 73 柏叶 61 60 56 九节茶 63 61 70 救必应 40 36 32 2.2 HERBGAT模型在探究半夏治疗肺癌作用机制中的应用
为进一步评估HERBGAT模型的综合性能,本文在国医大师周仲瑛[24-25]的经典处方中选取治疗肺癌的关键药物半夏,利用HERBGAT模型挖掘出中药半夏的潜在靶点关联基因,并利用David生信平台,从生物学角度对其进行Disease富集分析、KEGG信号通路分析。最终将模型的预测结果与文献检索结果综合考量分析,从中药-疾病的角度,对半夏治疗肺癌的作用机制进行了深入挖掘探究。
使用“1.2.1”与“1.2.2”中的相关方法进行中药-基因-蛋白复杂网络构建,其中模型的阳性样本即中药关联基因从TCMSP数据库中提取,本研究以OB≥30%,DL≥0.18作为筛选条件,得到半夏的12种关键活性成分24-Ethylcholest-4-en-3-one、Cavidine、Baicalein、Baicalin、β-Sitosterol、Stigmasterol、Gondoic acid、Coniferin、10, 13-Eicosadienoic、12, 13-Epoxy-9-hydroxynonadeca-7, 10-dienoic acid、Cycloartenol、β-D-Ribofuranoside,通过PubChem库寻找到与其关联的靶点基因309个,将这些基因作为阳性样本。为得到模型的阴性样本,本文在目前已知的全体人类基因组中,删除NCG、COSMIC、OMIM、DigSee数据库和KEGG癌症信号通路中的所有致癌、致病基因。最终输入数据集包括309个阳性样本和6 290个阴性样本。将整合好的输入矩阵,与包含多组学特征的基因蛋白网络一起输入HERBGAT模型中,挖掘发现潜在的半夏治疗肺癌的Top 500关联基因。接下来,对潜在中药关联基因使用系统生物学方法进行分析,探究半夏在肺癌治疗中的作用靶点信息,为中药作用机制的研究提供新的思路和方法。
2.2.1 半夏潜在关联基因的疾病富集分析
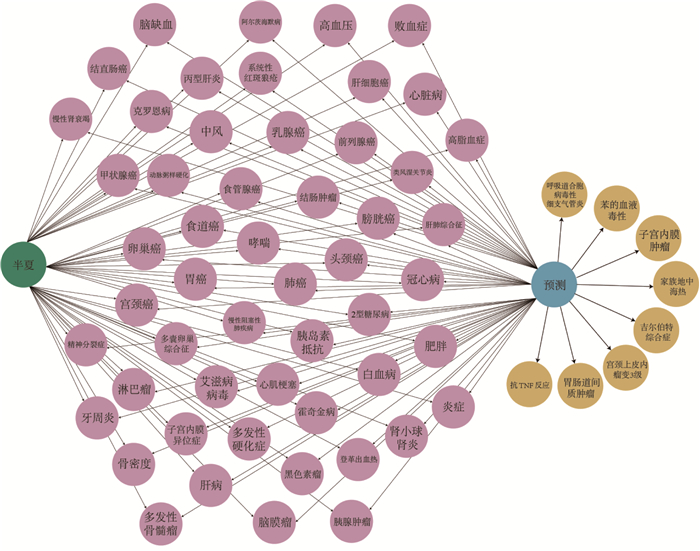
首先,对于图神经网络预测得到的半夏Top500关联基因,采用与“2.1”相同的验证方法,通过对中药关联基因进行疾病富集分析,发现半夏疾病富集分析结果在中医药文献中的检索命中率达到92%,这说明大部分半夏疾病富集分析结果都能得到现有的中医药文献验证。接下来,本文选择与半夏疾病富集分析结果相关的中医临床、中药药理研究文献作为考察范围,将其与半夏疾病富集分析结果进行关系分析,绘制半夏-疾病关联关系知识图谱,图 5展示了其中的部分内容,其中绿色节点半夏指向文献数据库中检索到的疾病,蓝色节点指向疾病富集分析得到的疾病,中间粉色节点为疾病富集分析在文献检索中命中的部分展示,黄色节点为仅预测得到的疾病。
从图 5可以观察到,半夏与哮喘相关(疾病富集分析结果中排名41,P=7.31E-15),且在中医临床用药中,半夏常被用作一味具有化痰止咳平喘功效的中药。现代药理学研究表明半夏[26]对治疗咳喘痰多、呕吐反胃、胸脘痞闷、瘰疬痰核等疾病具有显著疗效,这与知识图谱高度一致,验证了模型预测的准确性,进一步支持了半夏在处理哮喘等疾病方面的疗效。由中国医师协会中西医结合医师分会内分泌代谢病专业委员会制定的《2型糖尿病病证结合诊疗指南》[27]中指出,半夏是治疗湿热蕴结证、痰浊中阻证等肥胖型2型糖尿病的主治药物之一。半夏疾病富集分析结果中2型糖尿病的P值为1.20E-59,在所有预测疾病中排名第1,与2型糖尿病关联基因占比46%,也排名第1位。半夏疾病富集分析结果中肥胖(Obesity)排名第49(P=2.27E-13),排在与2型糖尿病相关的肝部疾病(P=1.92E-13)之后。现代药理学研究文献表明,半夏在抗肿瘤、抗菌、抗炎、抗癫痫、降血脂等方面表现出良好的药理活性[28-29]。半夏[30]是中医临床中固本解毒祛瘀法治疗非小细胞肺癌的重要药物之一,疾病富集分析结果中肺癌在所有预测疾病中排名第4(P=3.71E-42),且关联基因数量占比21%、排名第3。基于上述分析,半夏在肺癌等诸多疾病的治疗中,可以起到积极有效的治疗作用。且妊娠并发症疾病在疾病富集分析结果中排名66(P=1.03E-09),《本草纲目》[31]列出了八十多味妊娠期禁忌药物,半夏名列其中,古籍中对半夏妊娠毒性的记载进一步验证模型对中药性质的深入挖掘与准确分析。
2.2.2 半夏治疗肺癌作用机制分析
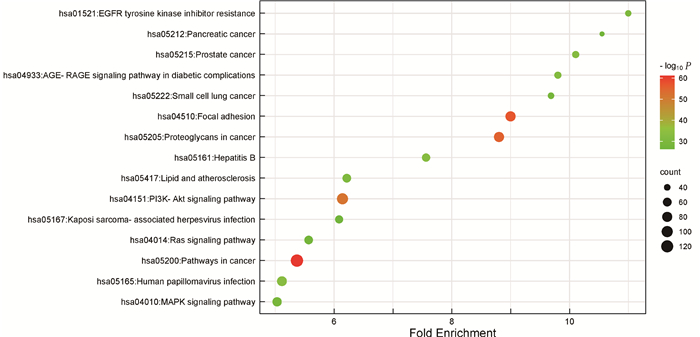
根据HERBGAT模型预测出的半夏的Top 500关联基因,通过KEGG平台,挖掘出半夏在相关肿瘤中可能调节的信号通路,依据Fold enrichment、Gene counts和P值进行分析,筛选出半夏Top15信号通路(图 6)。
通过研究发现,在GCN模型的基础上引入图注意力机制的方式,可以提高潜在癌症关联基因的预测性能。如EMOGI[13]算法以GCN方法为算法核心,在CPDB网络中的肺癌关联基因预测准确率为82%。而引入图注意力机制对其加以改进后,在CPDB网络中肺癌关联基因预测准确率可达92%。故本文使用改进后的EMOGI算法,针对肺鳞癌、肺腺癌与小细胞肺癌,进行了肺癌关联基因的预测。通过在DigSee、NCG等数据库中提取出已有的肺癌相关基因,经数据预处理后,得到肺癌输入特征矩阵,利用改进的EMOGI算法预测分析后,最终给出基因蛋白网络中未知基因与肺癌的关联置信度,排序后筛选出肺癌Top 500潜在关联基因。
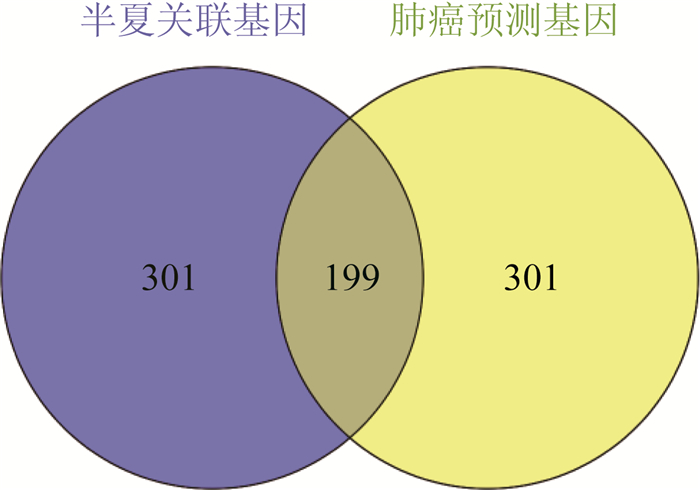
对HERBGAT算法预测出的Top 500潜在半夏关联基因与通过改进的EMOGI算法预测出的Top 500潜在肺癌关联基因进行交集运算,见图 7。通过交集运算结果,可发现半夏用于治疗肺癌的潜在关联基因199个,相关性排列详见图 3。其中包含ALK[32]、MET[33]等与肺癌密切相关的致病基因;BID[34]、FURIN[35]等参与细胞信息传导的G蛋白偶联受体基因(常见药物靶点基因)等靶点基因。接下来,对潜在关联基因集进行KEGG通路富集后,根据P值排名筛选出Top 5的信号通路,见表 3。本研究发现预测基因主要富集于两类信号通路,一类是在与肺癌或其他癌症关联性较大信号通路,另一类是参与癌症及其他疾病免疫的信号通路,这为未来的实验研究提供了数据基础和方向。
表 3 新预测的半夏治肺癌的潜在关联基因Top 5信号通路Table 3. New predicted top 5 signal pathways of potential associated genes for Pinellia ternata in treating lung cancer通路 基因数 百分比/% P T cell receptor signaling pathway 30 15.0 6.3E-23 Pathways in cancer 52 26.0 1.2E-20 PD-L1 expression and PD-1 checkpoint pathway in cancer 25 12.5 1.99E-20 Proteoglycans in cancer 33 16.5 3.3E-19 PI3K-Akt signaling pathway 40 20.0 1.3E-17 通过图 7,本文将半夏关联基因和肺癌预测基因交集运算获得预测的半夏治疗肺癌的潜在关联基因靶点199个。
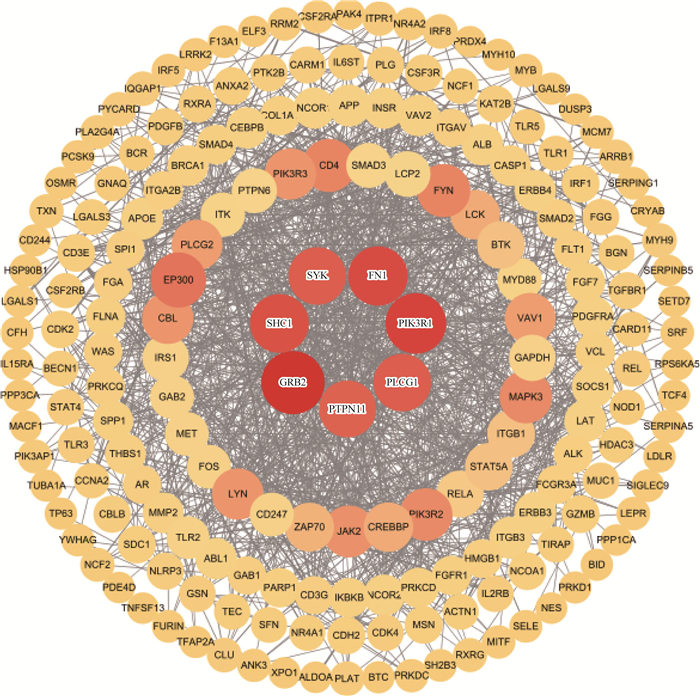
向STRING数据库中导入预测基因集,限定物种为Homo Sapiens,获得其PPI网络图。该网络图包括199个节点,2 419条高置信度边(Confidence>0.7)。导入Cytoscape3.10.0软件进行分析后展示,如图 8。其中节点代表靶点基因,节点形状越大、颜色越深代表Degree值越高。
接下来,本文对半夏关联基因Top 15信号通路和半夏治肺癌预测基因Top 5信号通路中部分共有信号通路及关键通路,进行了文献检索和分析,通过半夏治疗肺癌作用的信号通路,补充说明了半夏起效的作用机制。
(1) T cell receptor signaling pathway
T cell receptor signaling pathway是调控免疫系统对抗原做出响应的关键机制,能够确保免疫系统能够有效地识别、攻击和记忆病原体。其可以有效识别肿瘤细胞表面的异常抗原,引发免疫应答,导致肿瘤细胞的破坏和清除[36],同时其可以促使其他免疫细胞参与,共同协作清除感染的病原体。
(2) EGFR tyrosine kinase inhibitor resistance、PI3K-Akt signaling pathway、MAPK signaling pathway信号通路
肿瘤表皮生长因子受体(EGFR)是一个170 kDa的跨膜糖蛋白受体酪氨酸激酶,由表皮生长因子激活,影响细胞的生长和分化。EGFR基因族在肺癌中的表达率高达40%~80%,主要参与细胞增殖基因的调节、细胞凋亡基因的调控及参与肿瘤生长、转移、浸润相关基因的调控。陶兴宝[37]通过建立小鼠腹腔炎症模型和家兔眼刺激模型,采用不同浓度、不同pH的甘草汁、石灰水浸泡等多种方式,对半夏的中药炮制品法半夏进行了长期有效的对比实验,并结合半夏的毒性实验研究发现,法半夏中的有机酸类成分、生物碱类成分、氨基酸类等多种化学物质,以原型成分被吸收入血而发挥作用,抑制EGFR/MAPK/PI3K-Akt信号通路的激活[38],下调MUC5AC的表达,上调AQP5的表达,从而发挥化痰效应。这对肺癌并发症治疗起到中药积极作用,与模型预测结果吻合。
(3) Pathways in cancer、Proteoglycans in cancer信号通路
Pathways in cancer中整合了与多个癌症相关的信号通路,如黏附、凋亡、血管内皮生长因子、线粒体等信号通路,其中涉及癌细胞生存、增殖、生长等多个调控过程。半夏在药理作用性质中表现出明显的抗肿瘤作用,比如在肺癌治疗中,半夏总生物碱[39]可以通过抑制人肺癌细胞株A549增殖,起到肺癌的治疗作用。Proteoglycans in cancer信号通路主要包括蛋白多糖基因表达对癌症的影响,其中研究者曾从半夏多糖的抗肿瘤作用与其抗氧化作用进行相关性研究,发现半夏多糖不仅可以增强机体的免疫功能,提高免疫器官脾的质量,还能直接对肿瘤细胞进行杀伤,同时也可提高机体内酶的活力,清除多余的自由基[40]。
3. 讨论
本研究构建中药-基因-蛋白复杂网络,提出融合注意力机制的图神经网络模型HERBGAT,用于挖掘潜在的中药关联基因。为进一步提高模型的鲁棒性和泛化性,本研究经过多组训练数据的反复论证及多种方法相互比较、实验的方式,通过不断调整模型的图注意力卷积层数、隐藏层数、特征矩阵提取方式及数据处理通道数等方式,优化模型性能。本研究利用HERBGAT模型批量训练了99味中药,收集其结果数据,并将预测出的基因进行疾病富集分析,通过文献验证了分析结果。经验证本模型基于已有较少的中药靶点关联信息,可以有效找到潜在的中药关联基因,进而帮助科研人员从中药整体角度出发,探究中药与疾病靶点间作用机制。本研究最后展示了利用HERBGAT模型,探究半夏治肺癌作用机制中系统生物学信息挖掘的研究流程,并通过中医临床文献及现代药理学文献,对挖掘出的系统生物学信息进行验证。本研究为中药系统生物学信息挖掘方法的优化提供了新的思路与参考,为探究中药作用机制研究提供生信数据基础及未来实验方向。
在后续研究中,会进一步扩大数据集选取范围,通过多个公开平台数据的相互补充,提高挖掘结果的真实性、广泛性,并通过纳入更多基因生物学特征的方式,增加网络包含的可利用信息,以此提高信息挖掘的效能。
-
表 1 3种算法对99种中药训练出的性能均值
Table 1 Performance averages of three algorithms trained on 99 traditional herbal medicines
算法 ACC AUC AUPR HERBGAT 0.94 0.83 0.52 HERBGCN 0.89 0.70 0.23 PageRank 0.49 0.71 0.12 表 2 预测的Top100疾病在文献数据库中的检索命中率
Table 2 Retrieval hit rates of the predicted top 100 diseases in literature databases
中药 HERBGAT_ACC/
%HERBGCN_ACC/
%PageRank_ACC/
%姜 91 93 88 桔梗 89 91 84 白鲜皮 85 82 78 京大戟 68 63 61 橘红 81 81 73 柏叶 61 60 56 九节茶 63 61 70 救必应 40 36 32 表 3 新预测的半夏治肺癌的潜在关联基因Top 5信号通路
Table 3 New predicted top 5 signal pathways of potential associated genes for Pinellia ternata in treating lung cancer
通路 基因数 百分比/% P T cell receptor signaling pathway 30 15.0 6.3E-23 Pathways in cancer 52 26.0 1.2E-20 PD-L1 expression and PD-1 checkpoint pathway in cancer 25 12.5 1.99E-20 Proteoglycans in cancer 33 16.5 3.3E-19 PI3K-Akt signaling pathway 40 20.0 1.3E-17 -
[1] 潘锋. 中医药基础研究重在阐明科学内涵[N]. 科学时报, 2010-09-13(B2). PAN F. The fundamental research of traditional Chinese medicine focuses on clarifying its scientific connotations[N]. Science Times, 2010-09-13(B2).
[2] 程建超, 童佳兵, 朱洁, 等. 基于792份住院病历探讨中医药治疗肺癌的处方规律[J]. 时珍国医国药, 2020, 31(9): 2278-2280. CHENG J C, TONG J B, ZHU J, et al. Based on 792 inpatient medical records, this paper discusses the prescription law of traditional Chinese medicine in treating lung cancer[J]. Lishizhen Med Mater Med Res, 2020, 31(9): 2278-2280.
[3] GAN X, SHU Z X, WANG X Y, et al. Network medicine framework reveals generic herb-symptom effectiveness of traditional Chinese medicine[J]. Sci Adv, 2023, 9(43): eadh0215. DOI: 10.1126/sciadv.adh0215
[4] MASTROPIETRO A, PASCULLI G, BAJORATH J. Learning characteristics of graph neural networks predicting protein-ligand affinities[J]. Nat Mach Intell, 2023, 5: 1427-1436. DOI: 10.1038/s42256-023-00756-9
[5] RU J L, LI P, WANG J N, et al. TCMSP: A database of systems pharmacology for drug discovery from herbal medicines[J]. J Cheminform, 2014, 6: 13. DOI: 10.1186/1758-2946-6-13
[6] DOMS A, SCHROEDER M. GoPubMed: Exploring PubMed with the gene ontology[J]. Nucleic Acids Res, 2005, 33: W783-W786. DOI: 10.1093/nar/gki470
[7] LIU J F, LICHTENBERG T, HOADLEY K A, et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics[J]. Cell, 2018, 173(2): 400-416. DOI: 10.1016/j.cell.2018.02.052
[8] SEAL R L, BRASCHI B, GRAY K, et al. Genenames. org: The HGNC resources in 2023[J]. Nucleic Acids Res, 2023, 51: D1003-D1009. DOI: 10.1093/nar/gkac888
[9] KIM S, CHEN J, CHENG T J, et al. PubChem 2019 update: Improved access to chemical data[J]. Nucleic Acids Res, 2019, 47: D1102-D1109. DOI: 10.1093/nar/gky1033
[10] YAN D Y, ZHENG G H, WANG C C, et al. HIT 2.0: An enhanced platform for Herbal Ingredients'Targets[J]. Nucleic Acids Res, 2022, 50: D1238-D1243. DOI: 10.1093/nar/gkab1011
[11] KAMBUROV A, WIERLING C, LEHRACH H, et al. ConsensusPathDB: A database for integrating human functional interaction networks[J]. Nucleic Acids Res, 2009, 37: D623-D628. DOI: 10.1093/nar/gkn698
[12] JOHNSON W E, LI C, RABINOVIC A. Adjusting batch effects in microarray expression data using empirical Bayes methods[J]. Biostatistics, 2007, 8(1): 118-127. DOI: 10.1093/biostatistics/kxj037
[13] KILLOCK D. Genetics: HotNet2-see the wood for the trees[J]. Nat Rev Clin Oncol, 2015, 12(2): 66.
[14] WATTS D J, STROGATZ S H. Collective dynamics of small-world networks[J]. Nature, 1998, 393(6684): 440-442. DOI: 10.1038/30918
[15] BARABASI A L, ALBERT R. Emergence of scaling in random networks[J]. Science, 1999, 286(5439): 509-512. DOI: 10.1126/science.286.5439.509
[16] GONZALEZ-GOMARIZ J, SERRANO G, TILVE-ALVAREZ C M, et al. UPEFinder: A bioinformatic tool for the study of uncharacterized proteins based on gene expression correlation and the PageRank algorithm[J]. J Proteome Res, 2020, 19(12): 4795-4807. DOI: 10.1021/acs.jproteome.0c00364
[17] GREENACRE M, GROENEN P J F, HASTIE T, et al. Principal component analysis[J]. Nat Rev Meth Prim, 2022, 2(1): 100. DOI: 10.1038/s43586-022-00184-w
[18] ZHANG T, LIN Y X, HE W M, et al. GCN-GENE A novel method for prediction of coronary heart disease-related genes[J]. Comput Biol Med, 2022, 150: 105918. DOI: 10.1016/j.compbiomed.2022.105918
[19] XIAO S X, WANG S P, DAI Y F, et al. Graph neural networks in node classification: Survey and evaluation[J]. Mach Vis Appl, 2021, 33(1): 4.
[20] VELICKOVIC P, CUCURULL G, CASANOVA A, et al. Graph attention networks[J]. stat, 2017, 1050(20): 10-48550.
[21] JIN W, QU M, JIN X S, et al. Recurrent event network: Autoregressive structure inference over temporal knowledge graphs[EB/OL]. (2019-04-11)[2024-03-05]. https://arxiv.org/abs/1904.05530v4.
[22] HUANG D W, SHERMAN B T, TAN Q N, et al. DAVID Bioinformatics Resources: Expanded annotation database and novel algorithms to better extract biology from large gene lists[J]. Nucleic Acids Res, 2007, 35: W169-W175. DOI: 10.1093/nar/gkm415
[23] 李弘. 医学主题词的选取[J]. 中国病理生理杂志, 2000, 16(5): 478-480. LI H. Selection of medical subject headings[J]. Chin J Pathophysiol, 2000, 16(5): 478-480.
[24] 周仲瑛, 吴勉华, 周学平, 等. 中医辨治肿瘤十法[J]. 南京中医药大学学报, 2018, 34(6): 541-548. https://xb.njucm.edu.cn/article/id/zr20180601 ZHOU Z Y, WU M H, ZHOU X P, et al. Ten methods of tumors from TCM syndrome differentiation[J]. J Nanjing Univ Tradit Chin Med, 2018, 34(6): 541-548. https://xb.njucm.edu.cn/article/id/zr20180601
[25] 周计春, 邢风举, 颜新. 国医大师周仲瑛教授治疗癌毒五法及辨病应用经验[J]. 中华中医药杂志, 2014, 29(4): 1112-1114. ZHOU J C, XING F J, YAN X. Traditional Chinese medicine master ZHOU Zhong-Ying's five kinds of methods in anticancer and toxin expelling and his experience in disease differentiation[J]. China J Tradit Chin Med Pharm, 2014, 29(4): 1112-1114.
[26] 左军, 牟景光, 胡晓阳. 半夏化学成分及现代药理作用研究进展[J]. 辽宁中医药大学学报, 2019, 21(9): 26-29. ZUO J, MOU J G, HU X Y. Research progress in the chemical constituents and modern pharmacological effects of Pinellia ternata[J]. J Liaoning Univ Tradit Chin Med, 2019, 21(9): 26-29.
[27] 庞国明, 倪青, 张芳. 2型糖尿病病证结合诊疗指南[J]. 中医杂志, 2021, 62(4): 361-368. PANG G M, NI Q, ZHANG F. Guideline for diagnosis and treatment of type Ⅱ diabetes based on syndrome differentiation combining with disease differentiation[J]. J Tradit Chin Med, 2021, 62(4): 361-368.
[28] 张明发, 沈雅琴. 半夏提取物抗菌抗炎及其抗肿瘤药理作用研究进展[J]. 抗感染药学, 2017, 14(6): 1089-1094. ZHANG M F, SHEN Y Q. Research progresses of pharmacological actions in antimicrobial, anti-inflammation and antitumor of extract from pinelliae rhizoma[J]. Anti Infect Pharm, 2017, 14(6): 1089-1094.
[29] 张明发, 沈雅琴. 半夏及其炮制品对神经和循环系统的药理作用研究进展[J]. 抗感染药学, 2017, 14(9): 1643-1648. ZHANG M F, SHEN Y Q. Research progress in pharmacologic effects of pinelliae rhizoma and its processed products in nervous and circulatory systems[J]. Anti Infect Pharm, 2017, 14(9): 1643-1648.
[30] 许斌, 李文婷, 李丽, 等. 固本解毒祛瘀法与化疗联合治疗晚期非小细胞肺癌的临床研究[J]. 中华中医药学刊, 2024, 42(10): 48-51. XU B, LI W T, LI L, et al. Clinical study on treatment of advanced non-small cell lung cancer by combination of consolidating root, removing toxin and dispelling blood stasis method and chemotherapy[J]. Chin Arch Tradit Chin Med, 2024, 42(10): 48-51.
[31] 王全权, 宗芳, 陈海林. 对半夏妊娠毒性的探讨[J]. 时珍国医国药, 2007, 18(2): 330-331. WANG Q Q, ZONG F, CHEN H L. Discussion on pregnancy toxicity of Pinellia ternata[J]. Lishizhen Med Mater Med Res, 2007, 18(2): 330-331.
[32] HALLBERG B, PALMER R H. The role of the ALK receptor in cancer biology[J]. Ann Oncol, 2016, 27(Suppl 3): iii4-iii15.
[33] GHERARDI E, BIRCHMEIER W, BIRCHMEIER C, et al. Targeting MET in cancer: Rationale and progress[J]. Nat Rev Cancer, 2012, 12(2): 89-103.
[34] ESPOSTI M D. The roles of Bid[J]. Apoptosis, 2002, 7: 433-440.
[35] THOMAS G. Furin at the cutting edge: From protein traffic to embryogenesis and disease[J]. Nat Rev Mol Cell Biol, 2002, 3(10): 753-766.
[36] GAUD G, LESOURNE R, LOVE P E. Regulatory mechanisms in T cell receptor signalling[J]. Nat Rev Immunol, 2018, 18(8): 485-497.
[37] 陶兴宝. 法半夏炮制解毒机制、化痰效应及相关物质基础研究[D]. 南京: 南京中医药大学, 2022. TAO X B. Study on detoxification mechanism, expectorant effect and related material basis of processed Pinellia ternata[D]. Nanjing: Nanjing University of Chinese Medicine, 2022.
[38] TAO X B, LIU H B, XIA J, et al. Processed product (Pinelliae Rhizoma Praeparatum) of Pinellia ternata (Thunb. ) Breit. Alleviates the allergic airway inflammation of cold phlegm via regulation of PKC/EGFR/MAPK/PI3K-AKT signaling pathway[J]. J Ethnopharmacol, 2022, 295: 115449.
[39] 周茜, 唐瑛, 孙欢, 等. 半夏总生物碱对人肺癌细胞增殖的抑制作用[J]. 药学实践杂志, 2013, 31(1): 38-41. ZHOU X, TANG Y, SUN H, et al. Proliferation inhibition of total alkaloids from Pinellia Ternata in human lung cancer cells[J]. J Pharm Pract, 2013, 31(1): 38-41.
[40] 陈益. 半夏多糖的结构与抗肿瘤活性研究[D]. 西安: 陕西师范大学, 2007. CHEN Y. Study on structure and anti-tumor activity of polysaccharide from Pinellia ternata[D]. Xi'an: Shaanxi Normal University, 2007.



 下载:
下载: